
Questa linea di ricerca mira a definire come le vie metaboliche e i comparti subcellulari agiscano in modo integrato sia per regolare la vita delle piante e la loro risposta agli stress climatici, sia per determinare l’accumulo di sostanze utili per l’alimentazione e la salute umana. Sono parte di queste indagini: l’identificazione dell’espressione di geni coinvolti nella determinazione della struttura, del traffico e del metabolismo cellulare e della risposta a stimoli esterni; la caratterizzazione del ruolo funzionale e fisiologico delle proteine codificate. Queste analisi vengono condotte attraverso approcci sperimentali che utilizzano modelli in vivo, in vitro ed in silico.


La fotosintesi è il processo mediante il quale la luce solare è convertita in potenziale chimico, utilizzabile dagli organismi, e rappresenta pertantp il sito primario si produzione energetica nella biosfera. In condizioni ottimali le relazioni di foto-conversione procedono con una resa quantica estremamente elevata (80-99%). Lo studio di questi sistemi biologici può quindi rappresentate un paradigma utile per l’implementazione di materiali e strategie che li imitino per applicazioni fotovoltaiche e fotocatalitiche. D’altro canto, in condizioni naturali, a causa di variabilità e fluttuazioni ambientali, la resa fotosintetica è generalmente più bassa di quella ottimale e questo può portare a una riduzione della produttività, soprattutto per quanto riguarda le colture agricole. Pertanto, lo studio dei processi, come la raccolta della luce e il controllo dell’efficienza di raccolta, potrebbe portare ad un miglioramento della produttività delle colture ed essere quindi vantaggioso dal punto di vista sociale.
Il progetto “Enhancing Photosintesi” si propone di affrontare alcuni di questi aspetti, studiando sia i fattori che influenzano la produttività delle colture/piante, sia i loro meccanismi molecolari, sia i meccanismi fondamentali di conversione dei fotoni nei fotosistemi con l’obiettivo di trasferire alcune delle loro caratteristiche in sistemi biomimetici artificiali.
Il progetto prevede inoltre lo sviluppo di una piattaforma spettroscopica (ottica) per lo studio dei processi fotosintetici sia in(super)complessi clorofilla-proteina isolati, sia in sistemi fotosintetici intatti così come in sistemi sintetici artificiali (anche allo stato solido). La strumentazione coprirà oltre ad un ampio intervallo temporale (dai femto ai millisecondi) anche una ambia banda spettrale (dal vicino UV al vicino IR). Questa piattaforma sarà basata sia su strumentazioni già esistenti presso il CNR Milano e al PoliMI, le cui caratteristiche saranno sostanzialmente estese/implementate per renderle adatte allo studio di campioni molto diversificati e complicati da studiare a causa dell’elevata dispersione della luce che presentano. E’ fine del progetto rendere questo “hub” spettroscopico aperto che rappresenti una struttura di duratura che possa aggregare e promuovere la ricerca in fotosintesi, e su argomenti strettamente correlati, a livello regionale e possibilmente nazionale.


Per svolgere la propria funzione, tutte le proteine devono acquisire una forma ben specifica. Negli esseri viventi esiste in ogni cellula una macchina molto efficiente che trattiene qualsiasi proteina che non abbia completato la maturazione, rilasciandola soltanto quando ha raggiunto la forma e funzione corretta. Questo sistema di controllo di qualità è molto utile nelle persone sane; alcuni individui, però, nascono con delle mutazioni nel DNA che provocano un difetto in una proteina senza tuttavia abolirne totalmente la funzionalità. Se questo rigido controllo di qualità trattiene all’interno della cellula delle proteine difettose non mature possono esserci conseguenze gravi per l’organismo. In questo progetto studieremo dieci proteine difettose, scelte tra circa 6.000 proteine umane di cui si sa molto poco o addirittura nulla (“Proteine T-dark”). La nostra ipotesi è che rendendo il controllo di qualità meno severo, la proteina difettosa e la sua attività possano essere in parte recuperate. Seguiremo la traiettoria cellulare di tali proteine difettose in cellule private del sistema di controllo qualità e verificheremo se alcune saranno in grado di arrivare correttamente a destinazione. Se la nostra ipotesi fosse confermata, fornirebbe il razionale per un approccio farmacologico in grado di rendere il controllore della qualità più tollerante ai difetti di proteine mutate. I nostri risultati porranno le basi per una possibile terapia di una vasta gamma di malattie rare, accomunate dal fatto di essere provocate dall’estrema efficienza del controllo di qualità quando la proteina difettosa del paziente è ancora attiva.
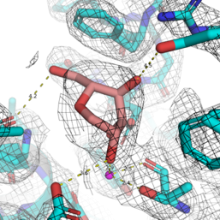
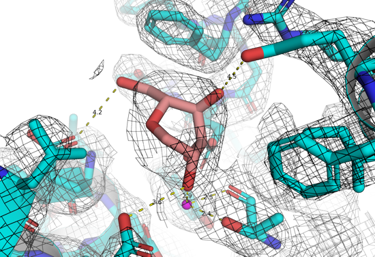
La degradazione associata al reticolo endoplasmatico (RE) è una strategia fondamentale della cellula eucariotica che le consente di degradare tutte le glicoproteine che non riescono a ripiegarsi correttamente, per evitare che un accumulo di glicoproteine mal ripiegate intasi il RE. La degradazione associata al reticolo endoplasmatico (ERAD) è avviata dal suo enzima di controllo, la mannosidasi di degradazione del reticolo endoplasmatico (EDEM), che è in grado di riconoscere qualsiasi glicoproteina mal ripiegata in modo terminale e di segnalarla per la degradazione. L’obiettivo specifico di questo progetto è identificare piccoli ligandi molecolari di HsEDEM3 che si leghino ai siti catalitici e/o allosterici e saggiare il loro potere inibitorio nella cellula. Questi ligandi costituiranno i punti di partenza per una futura chimica medicinale volta a sviluppare inibitori specifici per HsEDEM3. Attraverso lo screening Fragment Based Lead Discovery, ci proponiamo di identificare frammenti che si legano al sito catalitico di HsEDEM3 e composti che possono interferire con l’interazione del dominio catalitico con i domini C-terminali della stessa proteina. Il potenziale risultato a lungo termine di questo lavoro sarebbe un inibitore allosterico o un inibitore del sito catalitico di HsEDEM3 da usare in studi antivirali, antitumorali o per la terapia di alcune malattie rare. I nostri obiettivi sono: A: clonazione, espressione e purificazione di costrutti solubili del dominio catalitico GH47 dell’EDEM3 umana; B: cristallizzazione dei costrutti solubili del dominio catalitico GH47 dell’EDEM3 umana ottenuti al punto 1; C: Scoperta di ligandi dei costrutti solubili del dominio catalitico GH47 di EDEM3 umana (tramite Fragment-Based Lead Discovery con i cristalli ottenuti in 2.); D: Saggi di inibizione cellulare e in vitro di HsEDEM3 umano per verificare se i frammenti di ligandi di EDEM GH47 ottenuti in 3. sono inibitori di EDEM3


In un modello di Drosophila, è stato riscontrato che la promozione della degradazione associata all’ER (ERAD) attraverso l’aumento dei livelli di proteine α-mannosidasi-simili (EDEM) che condannano glycoproteine misfoldate a ERAD fornisce protezione contro la proteinopatia cronica dell’ER senza causare alcuna tossicità (10.1016/j.devcel.2017.05.019). Questo effetto protettivo è stato osservato senza influenzare la rete di espressione genica UPR. È interessante notare che in questi moscerini, con l’invecchiamento del cervello, l’attività ERAD è diminuita, ma l’upregolazione degli EDEM ha contrastato il declino comportamentale legato all’età e ha prolungato la durata della vita. In particolare, l’attività mannosidasica delle EDEM non era necessaria per questi effetti protettivi. Di conseguenza, il potenziamento della funzione degli EDEM nell’ERAD è promettente come potenziale bersaglio terapeutico per le malattie croniche. La nostra recente struttura Cryo-EM di un complesso EDEM:PDI suggerisce che l’attività redox del complesso è importante per la sua funzione: le glicoproteine clienti possono essere reclutate al complesso tramite disolfuri misti alla PDI. Utilizzeremo chimica delle proteine e biologia strutturale per confermare l’importanza dell’attività redox del checkpoint di misfolding ERAD, che a sua volta può spiegare il ruolo osservato di EDEM nell’invecchiamento, indipendentemente dalla sua attività di mannosidasi.


Il seme della pianta di granturco (Zea mays, detto anche mais) contiene zeine, proteine di stoccaggio del seme situate nel lume del Reticolo Endoplasmatico (ER) delle sue cellule. La principale proteina di stoccaggio del seme di mais, la Zea mays 27g-zeina (Zm27g-zeina), polimerizza nell’ER del seme di mais per formare grandi gabbie – che poi accumulano al loro interno altre proteine di stoccaggio del seme di mais, fornendo così un’impalcatura strutturale al corpo proteico (CP) che è la principale fonte di cibo del seme. L’N-terminus di Zm27g-zeina è necessario per la polimerizzazione che forma la gabbia PB – e il C-terminus della proteina si folda come un dominio di 2S-albumina – ma non sono disponibili strutture per l’intera proteina né per uno dei due domini separatamente. I modo in cui la Zm27g-zeina dà origine alla struttura molecolare della gabbia del CP è del tutto sconosciuto, comprese questioni quali la coesistenza intermolecolare Zm27g-zeina: interazioni covalenti e non covalenti tra monomeri di Zm27g-zeina formano la gabbia del CP; ci sono pori nella gabbia del CP e, in caso affermativo, di quali dimensioni; o l’orientamento relativo dei termini N- e C- nel contesto della gabbia del CP completamente formata (i termini N-/C- di Zm27g-zeina sono all’interno del CP, o all’esterno, o entrambi? ). Queste domande sulla gabbia di Zm27g-zeina del CP sono molto importanti per le applicazioni nei campi della scienza alimentare, dell’espressione di proteine ricombinanti, della farmacologia e delle nanotecnologie. Risponderemo a queste domande utilizzando la microscopia confocale, la cristallografia a raggi X e la crio-EM.


Il progetto vede l’ottimizzazione di strategie di bioincapsulamento di vaccini e farmaci utilizzando proteine di riserva dei semi di mais (zeine) come scaffold. Le zeine sono generalmente considerate sicure e possono anche fornire effetti adiuvanti per i vaccini.


Il tonoplasto (la membrana del vacuolo vegetale) svolge un ruolo centrale nella crescita della pianta e nell’omeostasi cellulare. Il network di ricerca VaTEP si è focalizzato sul ruolo di canali e trasportatori del tonoplasto per identificare le loro specifiche funzioni rispetto ai processi di sviluppo e crescita della pianta. Gli studi erano portati avanti in maniera interdisciplinare dai vari gruppi partecipanti e particolare attenzione era data alla formazione di giovani ricercatori.


Il progetto ad ampio spettro prevede:


Servizio di ingegneria proteica: produzione di costrutti ingegnerizzati (plasmidi) e verifica della sintesi e dell’accumulo, in cellule vegetali, di proteine chimeriche per utilizzi biotecnologici.


Il progetto BIO-ECO, coordinato dal Dipartimento di Scienze Bio Agroalimentari del CNR, vede coinvolto un team multidisciplinare di ricercatori provenienti da nove diversi Istituti distribuiti sull’intero territorio nazionale. Le attività di IBBA coprono:


Il calore e la siccità stressano le piante. Un ruolo importante nella resistenza a questo stress è svolto da un enzima chiamato Endoplasmic Reticulum Degradation Enhancing Mannosidase (“EDEM”). Questo enzima esiste in una forma resistente al calore in un fungo termofilo originariamente scoperto in un mucchio di letame di cavallo che fermentava al sole a circa 60-65 °C. La prima parte del progetto è volta a testare la resistenza allo stress di piante che esprimano quella EDEM di fungo termoresistente. Tale pianta transgenica potrebbe essere utile per estrarre metalli pesanti e/o indurre la degradazione di composti organici in terreni contaminati (fitorisanamento) oppure in agricoltura per avere aumentata produttività agricola in condizioni ostili (stress da caldo o da carenza di acqua durante fioritura e impollinazione). La seconda parte del progetto ambisce a ottenere un modello 3D di EDEM – per cristallografia a raggi X e/o criomicroscopia elettronica di trasmissione. Questo modello aiuterà a capire come funzioni EDEM e a progettare molecole per modularne l’attività catalitica. Oltre alla struttura, l’attività dell’enzima sarà caratterizzata in vitro, in cellula e in planta.


EU-IBISBA (Industrial Biotechnology Innovation and Synthetic Biology Accelerator) sostiene la ricerca nel campo delle biotecnologie industriali fornendo l’accesso a strutture di prima categoria per tutti i professionisti delle biotecnologie industriali. Nel 2018, questo concetto di infrastruttura di ricerca distribuita europea è stato inserito nella tabella di marcia del Forum strategico europeo sulle infrastrutture di ricerca. Una conseguenza immediata è stata l’ingresso del progetto nella fase di preparazione.
L’obiettivo del progetto di follow-up PREP-IBISBA, finanziato dall’UE, è quello di creare le condizioni affinché EU-IBISBA diventi un’infrastruttura di ricerca europea pienamente operativa nei prossimi anni. Il progetto aiuterà l’EU-IBISBA a definire un modello aziendale, a stabilire un piano finanziario a lungo termine e a identificare i quadri giuridici idonei a sostenerne il funzionamento

